近年来,结构DNA纳米技术的发展为在纳米尺度上精确设计构建各种复杂的纳米结构和器件提供了非常重要的策略和技术支持,最具代表性的策略有DNA origami和DNA brick等。特别是DNA brick策略提供了高度模块化的DNA链段设计,在构建无数复杂的形态各异的DNA纳米结构的同时,也为组装结构提供了尺寸无限拓展的可能。此外,所有的DNA链段都可以作为进一步功能化修饰且可寻址的位点,用于纳米材料的组装和超材料的制备。
但相对于目前应用较多的DNA origami策略,传统的DNA brick策略在组装可控性方面亟待提高。首先其组装过程受限于不可控的自发成核过程,导致组装的动力学较慢,组装路径随机不可控,此外由于成核需要克服较高的自由能势垒导致组装的热力学温度范围较高。因此,为了调控DNA brick结构的成核过程,从而在热力学和动力学上实现DNA brick结构的可控组装以及对组装路径的控制,北京化工大学张瑛洧(点击查看介绍)老师和美国Emory大学柯勇刚教授(点击查看介绍)合作,共同设计提出了“seeded-assembly”种晶调控组装策略。通过设计一条长的“种子”链段DNA,替代原DNA brick结构特殊位置的一些短链DNA,例如设计在二维结构的边缘位置或者三维结构的表面。该“种子”链段的引入为DNA brick结构组装带来了如下的优势:(1)降低了体系成核所需的自由能势垒,导致组装的优化温度范围大大增高,而较高的组装温度有利于减少组装过程中的错误,从而推进更多正确组装结构的形成;(2)加速了成核过程的动力学,从而完成组装的时间大大缩短;(3)“种子”链段在特殊边缘位置的设计可以提供一定的保护作用,有利于组装结构在较高温度范围稳定性的提高。研究者选择了4种有代表性的二维和三维结构作为模型,通过实验和计算拟合,成功地验证了“种子”链段带来的上述优势。此外,他们也通过AFM监控和表征了整个brick结构的组装路径,证实了组装过程的确是始于“种子”链段。这一新的“seeded-assembly”策略有助于加深对DNA brick结构组装过程的理解,并使研究者可以通过合理的设计来调控组装路径、组装动力学及热力学稳定性,为DNA brick结构未来的设计及应用提供了重要的指导意义。
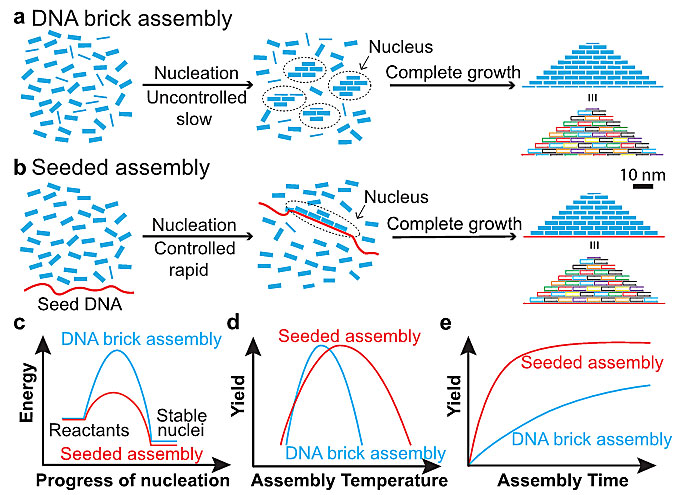
图1. 传统的DNA brick策略和“seeded-assembly”种晶调控组装策略在DNA 纳米结构的成核路径、组装热力学温度和动力学方面的对比。
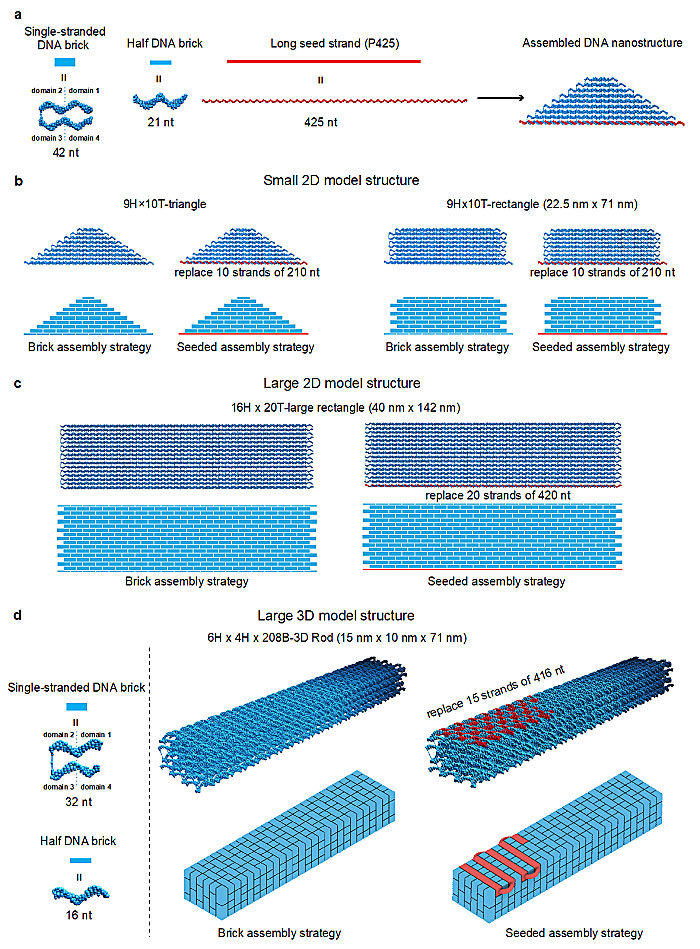
图2. 选择的四种代表性2D和3D模型结构,红色链代表种子链段。
该工作是北京化工大学张瑛洧老师课题组同美国Emory大学柯勇刚教授、英国剑桥大学Prof. Aleks Reinhardt 以及上海交通大学王鹏飞研究员等相关团队合作开展的,文章近期发表在Angew. Chem. Int. Ed.。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Programming the Nucleation of DNA Brick Self-Assembly with a Seeding Strand
Yingwei Zhang, Aleks Reinhardt, Pengfei Wang, Jie Song, Yonggang Ke
Angew. Chem. Int. Ed., 2020, DOI: 10.1002/anie.201915063
导师介绍
张瑛洧
https://www.x-mol.com/university/faculty/17340
柯勇刚
https://www.x-mol.com/university/faculty/117695
 0
0